加拿大多伦多大学的David J. Fleet和Ali Punjani合作开发了三维柔性精化(3DFlex)工具,可以通过冷冻电镜测定柔性蛋白的结构和运动。相关研究成果近日在线发表于《自然-方法学》。
据介绍,柔性大分子建模是单粒子冷冻电镜(cryo-EM)的首要挑战之一,有可能阐明结构生物学中的基本问题。
研究人员开发了3DFlex工具,这是一种基于运动的神经网络模型,用于冷冻电镜数据的连续分子异质性。3DFlex利用了这样一种知识,即蛋白质的构象变异通常是在空间上传输密度并倾向于保持局部几何形状的物理过程的结果。根据二维图像数据,3DFlex能够确定高分辨率的3D密度,并提供柔性蛋白质在其构象上运动的明确模型。
在实验中,对于大分子机器(三snRNP剪接体复合物、易位核糖体)和小的柔性蛋白质(TRPV1离子通道、αVβ8整合素、严重急性呼吸系统综合征冠状病毒2型刺突),3DFlex在学习非刚性分子运动的同时,能够解析移动二级结构元素的细节。
总之,3DFlex可以将3D密度分辨率提高到现有方法的极限之外,因为粒子图像在构象景观上贡献了相干信号。
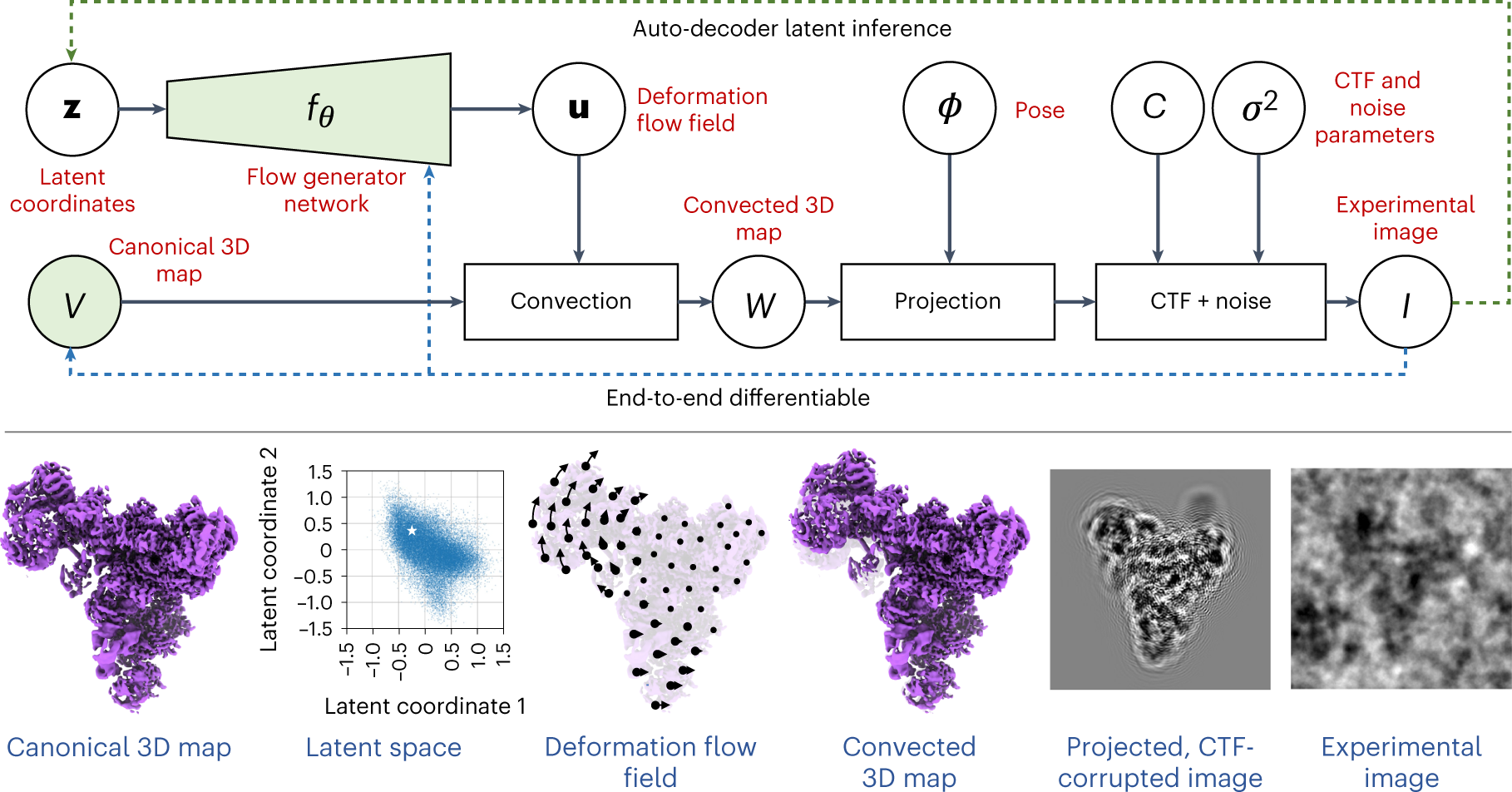
Punjani, A., Fleet, D.J. 3DFlex: determining structure and motion of flexible proteins from cryo-EM. Nat Methods (2023). 基于Creative Commons Attribution 4.0 International License再使用图片。